
新科技速遞
美國Covid-19病毒傳播初期,檢測技術出錯,抗疫受到挫折。美國軟件工業發達,不少行業處於醫療事業前端。
上周五,美國總統特朗普透露,Google母公司Alphabet附屬公司Verily Life Sciences(前稱Google Life Sciences)正開發網站,協助懷疑感染Covid-19冠狀病毒的患者自行分流測試。據Google傳訊部門表示,Verily處於開發初階段,計畫於三藩市灣區推出,期望擴展至更多地點。
美國總統特朗普透露超過1700名工程師,正參與有關Verily開發工程。Alphabet股價受刺激,一度上升超過9%。
Verily為Alphabet擁有的生命科學業務,據報網站讓用戶填寫問卷,自我檢測病癥,並獲悉從何可檢測,是否感染Covid-19冠狀病毒。網站亦介紹染感各種風險因素以及出現的病癥,用戶自行判斷有否屬於受感染風險,再決定到附近檢測中心,自行檢測病毒。
Google大舉投資人工智能,Verily可從網站和各種相關數據,預測長期病患,其至流行病風險;只要查詢身體狀況,再配合各種數據,就可推測出感染風險。Verily已在多國推動數據作診斷和醫療計畫,並取得成果。
人工智能開發新藥
Alphabet屬下另一家人工智能公司DeepMind,亦對於開發Covid-19病毒SARS-CoV-2解藥,可能有重大貢獻。2018年低,DeepMind以人工智能推出演算法AlphaFold,預測複雜3D形狀的蛋白質摺疊(Protein folding)競賽上獲冠軍。最近亦算出SARS-CoV-2的3D蛋白結構。
瞭解複雜3D形狀的蛋白質結構,被視為研發新藥的關鍵技術。較早前,美國德克薩斯大學奧斯丁分校團隊以「低溫電子顯微術」(Cryogenic electron microscopy,cryo-EM),快速成功拍攝SARS-CoV-2的「刺突蛋白」(Spike Protein),被視為開發病毒疫苗的突破。
SARS-CoV-2外圍有多枚刺突蛋白,外形看似冠狀,因此而被命名冠狀病毒。刺突蛋白藉結合人類細胞上的ACE2受體進行入侵,入侵方式與2002年出現SARS病毒一致;不過研究發現,SARS-CoV-2結合強度,竟比SARS病毒強出10至20倍,所以較SARS更易人傳人。
有關研究乃美國敏感及傳染病研究中心資助,2月19日的《自然》期刊上發表;又發現了現存針對SARS開發的三種抗體,均無法成功結合SARS-CoV-2刺突蛋白加以中和,如果開發病苗或以抗體為基礎的解藥,就必須另覓新蛋白結構。
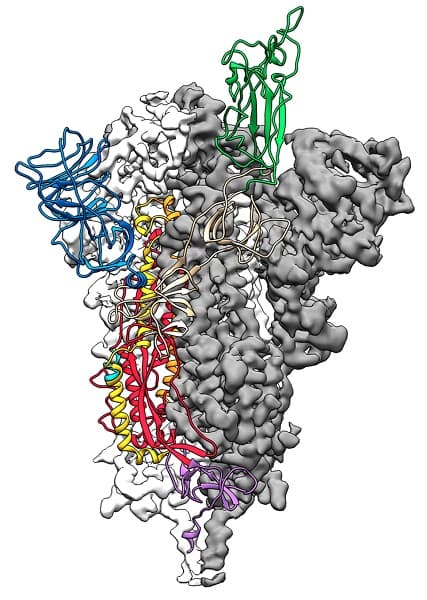
不少人以為,經過Covid-19冠狀病毒,不少國家均會投資cryo-EM,以期更快找出蛋白份子結構,加速新藥的開發。蛋白質是氨基酸通過肽鍵組成的線性鏈,摺疊成3D結構,結構取決於氨基酸序列和互相的物理作用,最重要是結構決定了蛋白質的生物學功能。
所以理解了病毒蛋白結構和功能,不單可理解病毒如何入侵人體細胞受體,以至如何作出抑制或中和病毒,但即使有低溫電子顯微術,人類理解的蛋白結構數據,仍然甚少。
美國國家科學基金會生物學部建立了蛋白質數據銀行(PDB,Protein Data Bank),支援全球上載3D形狀的蛋白結構資料處理,以加速開發新藥。
目前,以傳統「X射線晶體學」( X-ray crystallography)重建的蛋白份子結構,每年不足1萬,「X射線晶體學」非常耗時,往往是緩不濟急。cryo-EM可更快獲得影像,但以往影響模糊。近年在電腦幫助下大有改善。2017年, Richard Henderson就是憑 改善cryo-EM拍攝蛋白結構的清晰度,並加快拍 進程獲得了諾貝爾獎。
「X射線晶體學」極為耗時,cryo-EM安裝和操作昂貴,也不容易普及。德克薩斯大學短時期內,重建SARS-CoV-2冠狀病毒上刺突蛋白的結構,已經引起了哄動。不過決定整個結構仍然極為耗時,部分蛋白結構的來龍去脈,甚至無從稽考。
近期,科學家就利用了儲存蛋白質的氨基酸序列(Amino acid sequence),只要以前相同蛋白結構已經獲得實驗證明,就可以「模塊建模方式」(Template modelling),通過超級電腦以模擬計算,可估計出蛋白的結構。
以電腦預測的蛋白結構,一些特定蛋白質結構編碼後,以不同相似蛋白質結構,建立監督模型加以訓練,就可預測出結構。但是假如蛋白質結構從未重構過,舊的預測模式,就一籌莫展。
2018年底, DeepMind開發了AlphaFold演算法模型學習,並取得重大突破,竟能算出從未重構過的蛋白結構,稱為「自由建模方式」(Free modelling);第13屆全球蛋白質結構預測競賽(Critical Assessment of protein Structure Prediction,CASP)上,終於大放異彩。
Alphabet演算法凖確度驚人,43種蛋白質之中,準確預測其中25種,大幅領先第二名,只能凖確預測出其中3種蛋白結構。部分蛋白質結構生物學家甚至認為,AlphaFold演算法是研究蛋白結構最大突破之一。
DeepMind再利用了AlphaFold演算法,網上公佈了數個SARS-CoV-2蛋白結構的預測。DeepMind團隊強調,有關蛋白結構並未經實驗實證,不過AlphaFold演算法命中率,有關結構可助科學家研究,估計有關蛋白各種特性和功能,甚至配對以現存藥對付Covid-19。
去年,麻省理工學院公佈人工智能模型,可從氨基酸序列和蛋白結構,直接推斷蛋白的功能。類似技術應用研究疫苗或者抑制劑,均具重大意義;只要AlphaFold估算出結構,也能獲悉有關蛋白的功能。


